tidymodelを使ったサイトは意外と少なく、苦労したので記録として残しておきます。
※本記事は以前私が別サイトでまとめた以下の記事を追記・修正したものです。
tidymodel RandomForestによるクラス分類 https://qiita.com/nekobo/items/9637afaee5d5b00aafd5
パッケージのインストール
# rm(list = ls())
library(tidyverse)
library(dplyr)
library(ggplot2)
library(tidymodels)
library(skimr) # パッケージ名には「*r」が付くので注意
library(partykit) # 決定木の可視化
library(parallel) # rfの際の並列処理
library(withr) # シード値の固定 with_seed()
library(pROC) # ROC関連データの読み込み
url = "https://www.salesanalytics.co.jp/e4ye" # タイタニック
df = read.csv(url) %>% mutate(survived = as.factor(survived))前処理
エンコーディング
# sexを1/0に変換
df =
df %>%
mutate(sex =
case_when(
sex == "female" ~ "0",
sex == "male" ~ "1",
TRUE ~ as.character(sex)
)) %>%
mutate(sex = as.numeric(sex))
# one_hot_encoding
df =
recipe(survived ~ .,data = df) %>%
step_dummy(embarked) %>%
prep() %>%
bake(new_data = NULL) # 処理を適用した結果を返すbake():tibble型のデータを返す。new_data=NULLの場合、recipe()で指定されたデータが対象。
<step_関数の列指定に役立つヘルパ関数>
all_numeric() 数値型(integer型,double型)
all_nominal() 文字列型(character型,factor型)
all_numeric_predictors() 数値型(integer型,double型)で説明変数
all_nominal_predictors() 文字列型(character型,factor型)で説明変数
all_predictors() 説明変数
all_outcomes() 目的変数
start_with() 列名の接頭語で指定
contains() 指定した文字列が含まれる列を指定<all_outcomes()を使うときの注意点>
step_log(all_outcomes(),skip = TRUE)fit()はできるがpredict()をskip=TRUEの記載なしで実行するとエラーが発生。
恐らくpredictが目的変数を読み込まない仕組みになっており(=本来予測に要らない部分なので)、対象が見つからずエラーとなる。skip=TRUEとすることで、学習時のみstepが適用される。
欠損値処理
# 欠損値の確認
colSums(is.na(df))
# knn(k近傍法)で補完
df =
recipe(survived ~ .,data = df) %>%
step_impute_knn(all_numeric(),neighbors = 7) %>%
prep() %>%
bake(new_data = NULL)データの分割
# 層別抽出
ti_split =
initial_split(data = df, prop = 0.7,strata = survived)|>
with_seed(seed = 1234, code = _)
# データの取り出し
ti_train = training(ti_split)
ti_test = testing(ti_split)
# cross validation
ti_cv =
vfold_cv(ti_train, v = 5)|>
with_seed(seed = 1234, code = _) 乱数生成の際のシード値を固定するwith_seed() は %>% ではなく |> を使う必要があります。vfold_cv()で分割後はanalysis(df_cv[1]),assessment(df_cv[1])で分析セット、検証セットの確認ができます。
ハイパーパラメータチューニング
チューニング用のワークフロー作成まで
# レシピ
ti_rec = recipe(survived ~ .,data = ti_train)
# モデルスペック
rf_spec =
rand_forest(mtry = tune(), min_n = tune(), trees = tune()) %>%
set_mode('classification') %>%
set_engine('ranger') %>%
set_args( # engine特有の引数の設定
importance = "impurity", # ジニ係数,defaultだと変数重要度は返らない
num.threads = parallel::detectCores(), # 実行環境におけるコア数を抽出し、並列処理
probability = TRUE) # TRUE = 確率を返す
# チューニング用ワークフローの作成
rf_wf =
workflow() %>%
add_recipe(ti_rec) %>% # レシピの設定
add_model(rf_spec) # モデルスペックの設定tune()で設定した箇所が探索したいパラメータです。
# モデル関数の引数など詳細確認
rand_forest
# モデル関数に使えるエンジン(パッケージ)の確認
show_engines("rand_forest")グリッドサーチの設定~実行まで
# グリッドサーチ範囲の設定
rf_param =
list(trees(range = c(100, 500)), # 試す決定木の数,rangeで範囲の手動設定可
min_n(range = c(5, 50)), # 各最終ノードにおける最小データ件数
mtry() %>% # モデルに採用する変数の数
finalize(ti_train %>% select(-survived))
) %>%
parameters()
# グリッドサーチ範囲の確認
rf_param %>% extract_parameter_dials("trees")
# グリッドサーチの値の組み合わせの決定
rf_grid_value =
rf_param %>%
grid_random(size = 5) # 本来は200などもっと大きい値にする
# グリッドサーチの実行
rf_grid =
rf_wf %>%
tune_grid(resamples = ti_cv, # 分割データを読み込ませる
grid = rf_grid_value, # グリッドデータを読み込ませる
control = control_grid(save_pred = TRUE), # オプション
metrics = metric_set(roc_auc)) |> # 評価指標
with_seed(seed = 1234, code = _)finalize(関数,df) で可変の範囲指定が可能。
グリッドサーチの結果の確認
# モデルの精度が良い順に結果をdfで取得
rf_grid_best = rf_grid %>% show_best()
# 以下の書き方でもほぼ同義
rf_grid_df =
rf_grid %>%
collect_metrics()
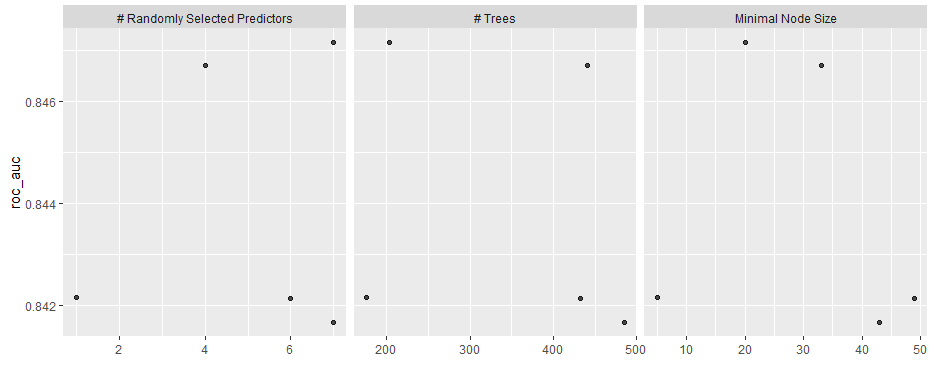
# 探索時の評価指標の値を可視化する
autoplot(rf_grid)
グリッドーサーチ結果の反映
# モデルスペックの再設定
rf_best_spec =
rand_forest(mtry = rf_grid_best$mtry[1],
min_n = rf_grid_best$min_n[1],
trees = rf_grid_best$trees[1]) %>%
set_mode('classification') %>%
set_engine('ranger') %>%
set_args(
importance = "impurity",
num.threads = parallel::detectCores(),
probability = TRUE)
# ワークフローの更新
rf_wf =
rf_wf %>%
update_model(rf_best_spec) # update_recipe()でレシピの更新<パターンa>学習と予測を同時に行なう last_fit
学習+予測
# splitデータを対象とする
rf_last_res =
rf_wf %>% # ワークフロー
last_fit(split = ti_split) |> # train/testを分割したデータ
with_seed(seed = 1234, code = _) last_fit()はtrainingデータで学習を実行し、さらにtestに対する予測も行う
評価と予測値を確認する
# テスト用データでの評価
rf_perf = rf_last_res %>% collect_metrics()
# テスト用データでの予測値(tibble型)
rf_res = rf_last_res %>% collect_predictions()<パターンb>学習と予測を別々に行う fit predict
学習・予測
# 学習
rf_fit =
rf_wf %>% # ワークフロー
fit(data = ti_train) |>
with_seed(seed = 1234, code = _) #乱数種種の固定
# 予測(tibble型)
rf_class_pred =
rf_fit %>% # 学習済みモデル
predict(new_data = ti_test)
# predict(…,type = "prob")を指定しなかった場合、
# 予測クラス.pred_classが返される(閾値不明)。
# [tips] predict()とaugment()
# 前者は「予測値」のみ、後者は「元のdf+結果列」を返す
# ただしaugment()の場合なぜか確率ではなく予測クラスが返された(要調査)精度検証
本章はtibble型データ全般に使用可.
指標の一括検証・混同行列
# 予測値を付与
ti_test_add_classpred = cbind(ti_test,rf_class_pred)
# 任意の指標一覧をmetric_set()で指定
ti_metrics = metric_set(accuracy,
sensitivity,
specificity,
precision,
recall,
f_meas)
# 真値と予測値の列を指定し、指標を一括計算
ti_test_add_classpred %>%
ti_metrics(truth = survived,
estimate = .pred_class) # factor型
# 真値と予測値の列を指定し、混同行列を計算
ti_test_add_classpred %>%
conf_mat(truth = survived,
estimate = .pred_class) # factor型一括計算の結果
ROC
# 確率型で予測値を出力・付与
rf_prob_pred =
rf_fit %>%
predict(new_data = ti_test,type = "prob")
ti_test_add_predprob = cbind(ti_test,rf_prob_pred)
# ROCオブジェクトの生成
ti_ROC = roc(survived ~ .pred_1,
data = ti_test_add_predprob,
ci = TRUE)
# ROC曲線の可視化
ggroc(ti_ROC)
# ROC上の最適解
coords(ti_ROC, "best")[1]
# 予測クラス列の追加
confusion_df = ti_test_add_predprob %>%
mutate(pred_class = if_else(.pred_1>coords(ti_ROC, "best")[1],1,0)) %>%
mutate(pred_class = as.factor(pred_class))
# 以降のステップは「指標の一括検証・混同行列」を参考変数重要度
# 変数重要度の取得
rf_importance =
rf_fit %>%
pluck("fit", "fit", "fit") %>%
importance()
# 変数重要度の出力
feature_importans_df =
tibble(feature = names(rf_importance),
importance = rf_importance)
# 可視化
feature_importans_df %>%
slice_max(importance, n = 50) %>%
ggplot(aes(y = reorder(feature, importance), x = importance)) +
geom_col()
参考文献
Rユーザのためのtidymodels[実践]入門 〜モダンな統計・機械学習モデリングの世界:書籍案内|技術評論社
tidymodels講座- データサイエンスの道標
Tidymodels使用時のオブジェクト(変数)命名規則【2022年度版】
Tidymodelsをシンプルに使う
Rで決定木分析(rpartによるCARTとrangerによるランダムフォレスト)


コメント